TỔNG HỢP PRIMER - PROBE
Mẫu dò là một phân tử acid nucleic (ADN sợi đơn hoặc ARN sợi đơn) có ái lực mạnh với đích đặc hiệu (trình tự ADN hoặc ARN). Mẫu dò và trình tự base đích bổ sung cho nhau, nhưng tùy thuộc điều kiện, chúng không nhất thiết phải bổ sung hoàn toàn. Phân tử lai (kết hợp mẫu dò – đích) có thể được phát hiện khi một hệ thống xác định và đánh dấu thích hợp được sử dụng. Các mẫu dò gen thường được áp dụng trong nhiều kỹ thuật blotting và các kỹ thuật lai tại chỗ (in situ techniques) nhằm mục đích xác định các trình tự acid nucleic. Trong y học, chúng có thể giúp định danh vi sinh vật và chẩn đoán các bệnh di truyền, bệnh truyền nhiễm và nhiều dạng bệnh khác.
2. Thiết kế mẫu dò
Thiết kế mẫu dò phụ thuộc vào yêu cầu sử dụng mẫu dò gen hay mẫu dò oligonucleotide.
2.1. Mẫu dò gen (Gene Probes)
Mẫu dò gen thường dài hơn 500 base và chứa toàn bộ (hay phần lớn) trình tự gen đích.
Chúng có thể được tạo ra bằng hai cách:
- Trong cách thứ nhất, các mẫu dò nhân bản (Cloned probes) được sử dụng khi có sẵn một bản nhân dòng đặc hiệu (specific clone) hay khi trình tự ADN chưa biết cần nhân dòng trước để lập bản đồ và giải trình tự. Gen đích được cắt bằng enzyme giới hạn và thu lại từ gel agarose. Trường hợp vector không tương đồng (homology), phản ứng cắt không cần thiết tiến hành. Phản ứng chuỗi polymerase (PCR) với khả năng khuếch đại ADN đồng thời đánh đấu các nucleotide, sử dụng khuôn là các ADN plasmid hay ADN nhiễm sắc thể, là quy trình sản xuất mẫu dò hiệu quả.
- Cách thiết kế thứ hai được tiến hành khi có toàn bộ trình tự gen từ các nguồn dữ liệu như GenBank, EMBL, DDBJ. Dựa vào trình tự tìm được, có thể dễ dàng thiết kế mồi (primer) để khuếch đại toàn bộ gen hoặc các đoạn gen bằng phản ứng PCR. Điều này giúp tiết kiệm nhiều thời gian vì bỏ qua bước cắt bằng enzyme giới hạn, bước điện di và bước rửa giải ADN từ vector. Tuy nhiên, nếu PCR cho sản phẩm chứa các băng không đặc hiệu, cần thực hiện tinh sạch các băng sử dụng làm mẫu dò để đảm bảo tính chính xác trong các thí nghiệm sau.
2.2. Mẫu dò oligonucleotide (Oligonucleotide Probes)
Mẫu dò oligonucleotide có đích là các trình tự đặc hiệu bên trong gen. Kích thước phổ biến của loại mẫu dò này là 18 – 30 base. Tuy vậy, các thiết bị tổng hợp hiện nay cho phép thiết kế hiệu quả các mẫu dò chứa ít nhất 100 base. Mẫu dò oligonucleotide gắn chính xác với trình tự đích. Chiều dài của chúng phù hợp cho phản ứng lai trong các điều kiện ngặt nghèo, giúp xác định ADN với những khác biệt rất nhỏ trong trình tự. Quá trình lựa chọn mẫu dò oligonucleotide có thể thực hiện sử dụng từ những trình tự gen đã biết, theo những bước sau:
- Chiều dài mẫu dò: 18 – 50 base. Kích thước dài hơn sẽ khiến thời gian lai lâu hơn và tỉ suất tổng hợp thấp, kích thước ngắn hơn sẽ làm giảm độ đặc hiệu.
- Thành phần G – C nên từ 40 – 60%. Nguy cơ lai không đặc hiệu tăng khi tỉ lệ G – C nằm ngoài khoảng trên.
- Chắc chắn rằng không có các vùng bổ sung bên trong mẫu dò vì chúng có thể tạo cấu trúc “kẹp tóc” (hair-pin) và ngăn cản quá trình lai.
- Tránh các trình tự chứa các đoạn nucleotide đơn liên tiếp (ví dụ như AAAA).
- Một khi trình tự đạt những yêu cầu trên, vẫn cần thiết phân tích trình tự trên máy tính. Nên so sánh trình tự mẫu dò với các vùng trình tự nguồn hay hệ gen nguồn cũng như so sánh với các trình tự bổ sung của các trình tự nguồn đó. Nếu độ tương đồng với các vùng (không phải đích) > 70% hoặc >=8 base trong 1 hàng được quan sát, mẫu dò đó không đủ điều kiện sử dụng.
Tuy nhiên, để xác định điều kiện lai tối ưu, nên tiến hành lai mẫu dò với các acid nucleic đích đặc hiệu và không đặc hiệu trong phạm vi biến đổi lớn về điều kiện lai.
Quy trình tương tự được áp dụng thiết kế mồi xuôi và mồi ngược dùng trong phản ứng PCR. Chú ý rằng, trừ vùng cần khuếch đại, đầu 3’ của mồi xuôi và mồi ngược không tương đồng với các đoạn ADN khuôn khác.
3. Đánh dấu mẫu dò
3.1 Các kiểu đánh dấu
3.1.1. Đánh dấu phóng xạ (Radioactive Labels)
Mẫu dò acid nucleic có thể được đánh dấu bằng các đồng phân phóng xạ (32P, 35S, 125I, 3H) và xác định bằng ảnh X– ray hay hệ thống Geiger– Muller. Trước đây, mẫu dò đánh dấu phóng xạ được sử dụng rất phổ biến, nhưng với những lo ngại về tính an toàn cũng như giá cả và vấn đề chất thải phóng xạ, ngày nay, người ta ít sử dụng chúng. Đây là loại mẫu dò có độ nhạy và độ phân giải cao nhất. Chỉ một lượng nhỏ sản phẩm lai mẫu dò– đích cũng có thể được phát hiện, ví dụ: mẫu dò đánh dấu 32P có khả năng xác định các bản sao của gen trong 0.5 µg ADN. Keller and Manak đã giải thích như sau:
- 32P có hoạt động đặc hiệu mạnh nhất.
- 32P phát hạt β có năng lượng cao.
Mặc dù mẫu dò loại này giúp phát hiện lượng nhỏ ADN đích (< 1pg), nhược điểm của chúng là thời gian bán hủy ngắn (chỉ sử dụng trong vòng 1 tuần sau khi chuẩn bị) và không thể sử dụng cho kỹ thuật hình ảnh độ phân giải cao. So với 32P, năng lượng thấp và thời gian bán hủy dài khiến đồng phân 35S ổn định và ít đặc hiệu hơn. Mẫu dò đánh dấu 35S dù có độ nhạy thấp hơn nhưng cho hình ảnh X – ray với độ phân giải cao hơn và đặc biệt thích hợp sử dụng trong kỹ thuật lai tại chỗ. Một ưu điểm khác của nucleotide đánh dấu 35S là ít độc hơn so với 32P. Các hạt b năng lượng thấp gần như không thâm nhập vào lớp da chết phía trên và dễ dàng được thu lại bằng các ống, lọ thí nghiệm. Tương tự, mẫu dò đánh dấu 3H đã và đang được sử dụng trong lai tại chỗ vì chúng phát ra các hạt β năng lượng thấp, kết quả là độ tương phản đạt cực đại và tín hiệu nhiễu thấp, chúng cũng có thời gian bán hủy dài nhất (12.3 năm). Việc sử dụng125I và 131I giảm kể từ những năm 1970 do sự sẵn có của mẫu dò triphosphate nucleoside đánh dấu 125I với tính đặc hiệu cao. 125I có năng lượng thấp hơn và thời gian bán hủy dài hơn (60 ngày) so với 131I, thường được sử dụng trong lai tại chỗ.
3.1.2. Đánh dấu không phóng xạ
So với đánh dấu phóng xạ, sử dụng các dấu chuẩn không phóng xạ có một vài ưu điểm:
- An toàn.
- Tính ổn định cao hơn.
- Phản ứng đánh dấu hiệu quả.
- Xác định tại chỗ.
- Cần ít thời gian để nhận biết tín hiệu.
Một số dấu chuẩn trong đánh dấu không phóng xạ:
- Biotin: Được phát hiện nhờ sử dụng avidin hoặc streptavidin. Streptavidin, avidin có ái lực mạnh mẽ với biotin. Vì enzyme báo cáo (reporter) không liên kết trực tiếp với mẫu dò mà gắn với nó thông qua một cầu nối (ví dụ: streptavidin–biotin) nên dạng xác định này được biết đến như một hệ thống gián tiếp. Thông thường, mẫu dò biotin hoạt động rất tốt nhưng vì biotin (vitamin H) là phân tử phổ biến trong mô động vật có vú và các mẫu dò biotin có xu hướng bám dính với những loại màng nylon nhất định nên tín hiệu nhiễu cao có thể xảy ra trong quá trình lai. Vấn đề này sẽ được giải quyết nhờ sử dụng dẫn xuất nucleotide, bao gồm: digoxigenen-11-UTP, -11-dUTP, -11-ddUTP, và biotin-11-dUTP hoặc biotin-14-dATP. Sau khi lai, nhận biết mẫu dò/ gen đích bằng kháng thể hoặc avidin, sau đó là phản ứng phát quang/ phản ứng màu được xúc tác bởi alkaline phosphatase hoặc peroxidase liên kết với kháng thể hoặc avidin.
- Enzyme: Phân tử enzyme (được gọi là nhóm báo cáo) gắn với mẫu dò và sự hiện diện của nó được xác định bởi phản ứng với cơ chất chuyển màu. Các enzyme thường dùng gồm alkaline phosphatase và horseradish peroxidase (HRP). Khi có mặt peroxide và peroxidase, chloronaphtol – một cơ chất của HRP tạo thành sản phẩm không tan màu tím. HRP cũng xúc tác quá trình oxi hóa của luminol – một phân tử phát quang.
- Chất phát quang hóa học (Chemiluminescence): Trong phương pháp này, các chất hóa học phát quang liên kết với mẫu dò được xác định bởi ánh sáng chúng phát ra. Mẫu dò phát quang hóa học (bao gồm cả mẫu dò enzyme nói trên) có thể tách khỏi màng, cho phép màng được lai nhiều lần mà độ phân giải không bị ảnh hưởng.
- Chất hóa học phát huỳnh quang (Fluorescence chemicals): gắn với mẫu dò huỳnh quang dưới tia UV. Mẫu dò loại này cực kỳ hữu dụng trong nghiên cứu mẫu tế bào hay mẫu vi sinh dưới kính hiển vi - kỹ thuật này có tên: lai huỳnh quang tại chỗ (fluorescent in situhybridization - FISH).

Hệ thống FISH
Một số vấn đề trong thiết kế mẫu dò cho FISH:
- Kháng thể: Một nhóm kháng nguyên kết hợp với mẫu dò, sự hiện diện của chúng được phát hiện bằng kháng thể đặc hiệu. Đồng thời, các kháng thể đơn dòng sẽ nhận biết phân tử lai ADN- ARN. Kháng thể tự đánh dấu hoặc có thể sử dụng enzyme.
- Hệ thống DIG: Đây là hệ thống hoàn thiện, tiện dụng và hiệu quả nhất trong đánh dấu và xác định ADN, ARN, oligonucleotide. Giống như biotin, digoxigenin (DIG) có thể gắn với cầu nối (linker) và nucleotide; các nucleotide được thay thể bởi DIG kết hợp mẫu dò acid nucleic bằng bất cứ phương pháp enzyme nào. So với mẫu dò biotin, các mẫu dò này tạo ít tín hiệu nhiễu hơn. Một phức hợp anti- digoxigenin kháng thể– alkaline phosphatase có khả năng gắn với mẫu dò đánh dấu DIG.
Tín hiệu được xác định với cơ chất tạo màu hay phát quang của alkaline phosphatase. Nếu sử dụng cơ chất tạo màu, tín hiệu hình thành trực tiếp trên màng. Trường hợp cơ chất phát quang, dùng phim X – ray để hiện tín hiệu.
Bảng 1. Các kiểu đánh dấu

3.2. Phương pháp đánh dấu
3.2.1. Phản ứng dịch chuyển điểm cắt (Nick Translation)
Phản ứng dịch chuyển điểm cắt là một phương pháp đánh dấu ADN, trong đó sử dụng các enzym DNase I tuyến tụy và Escherichia coli DNA polymerase I. Trong phản ứng, DNase I cắt mạch ADN ở một vài điểm (nick) và E. coli DNA polymerase I sẽ thêm nucleotide vào vị trí 3'-OH lộ ra ở các điểm cắt đó, đồng thời hoạt động 5’ - 3' exonuclease sẽ loại bỏ nucleotide ở đầu 5’. Nếu nucleotide đánh dấu đã có trong phản ứng, các nucleotide này sẽ tham gia vào mạch ADN. Đối với các ADN đánh dấu phóng xạ, [α-32P] dNTP sẽ được bổ sung. Đối với mẫu dò không phóng xạ, digoxigenin hoặc một đầu biotin gắn với một phân tử tương tự dNTP sẽ được sử dụng.
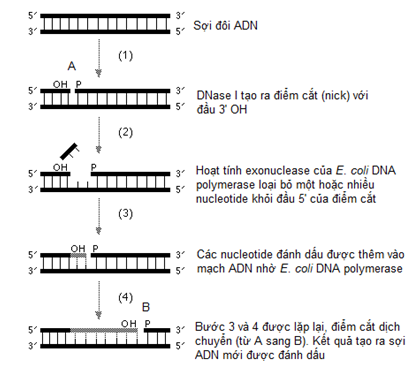
Phản ứng dịch chuyển điểm cắt (Nick Translation)
3.2.2. Đánh dấu bằng mồi ngẫu nhiên/ Kéo dài mồi (Random-Primed Labeling/ Primer Extension)
Mẫu dò gen (nhân dòng hay khuếch đại PCR), mẫu dò oligonucleotide có thể được đánh dấu bằng mồi ngẫu nhiên với các đồng phân phóng xạ và không phóng xạ (ví dụ DIG). Phân tử ADN sợi đôi trải qua quá trình biến tính và kết hợp với các đoạn mồi oligonucleotide ngẫu nhiên (6-mers). Các oligonucleotide này đóng vai trò như mồi cho 5' - 3' polymerase (đoạn Klenow). Mẫu dò đánh dấu được tổng hợp nhờ enzyme này khi trong môi trường có các nucleotide đánh dấu. Hình A thể hiện các bước trong đánh dấu DIG bằng mồi ngẫu nhiên.

Hình. (A). Đánh dấu DIG bằng mồi ngẫu nhiên. ADN sợi đôi bị biến tính và gắn mồi oligonucleotide ngẫu nhiên (6-mers); oligonucleotide này đóng vai trò như mồi cho đoạn Klenow của E. coli DNA polymerase I, enzyme sẽ tổng hợp mẫu dò đánh dấu DIG–dUTP. (B). Đánh dấu DIG bằng phản ứng PCR: DIG–dUTP tham gia mạch ADN trong quá trình khuếch đại khuôn ADN đích. Dấu hoa thị biểu hiện phân tử digoxigenin.
3.2.3. Đánh dấu DIG bằng phản ứng PCR (DIG–PCR Labeling)
Đây là một phương pháp tiên tiến đánh dấu mẫu dò gen với DIG sử dụng phản ứng PCR. Mẫu dò được khuếch đại sử dụng mồi phù hợp, tuy nhiên, hỗn hợp dNTP chứa ít dTTP vì DIG–dUTP cũng được thêm vào phản ứng này. Ưu điểm phương pháp này là gắn nhiều đuôi DIG dọc mạch ADN trong suốt quá trình khuếch đại. Hình B thể hiện các bước trong quá trình đánh dấu DIG bằng phản ứng PCR.
3.2.4. Đánh dấu photobiotin (Photobiotin Labeling)
Đánh dấu photobiotin là một phản ứng hóa học, không chỉ đơn giản là phản ứng enzyme. Biotin và DIG nối với một nhóm nitrophenyl azide. Nhóm này sẽ biến đổi bởi sự phát xạ với tia UV hoặc ánh sáng nhìn thấy, tạo phân tử nitrene hoạt động mạnh. Nhóm này có thể tạo liên kết cộng hóa trị vững chắc với ADN và ARN. Các vật liệu để đánh dấu photobiotin bền hơn enzyme trong phản ứng dịch chuyển điểm cắt hay kỹ thuật đánh dấu oligonucleotide và rẻ hơn các phương pháp khác. Tuy nhiên, lượng mẫu dò tạo ra lớn nhưng độ nhạy không cao.
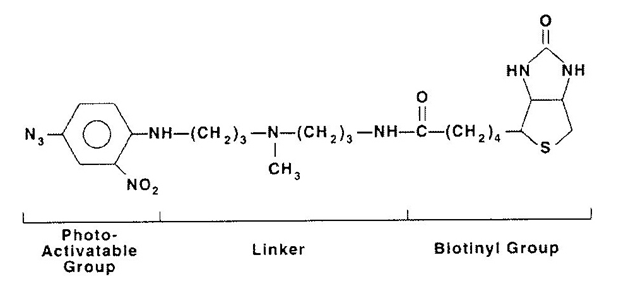
Photobiotin có công thức hóa học C23H35N9O45•C2H 4O2. Phân tử bao gồm một nhóm biotinyl, một nhóm nối (linker) cad một nhóm nitrophenyl azide.
3.2.5. Đánh dấu đuôi (End Labeling)
Phương pháp này chủ yếu dùng cho đánh dấu mẫu dò oligonucleotide.
Ba phương pháp đánh dấu mẫu dò oligonucleotide với digoxigenin:
- Đánh dấu đầu 3’ của một oligonucleotide kích thước 14 – 100 nucleotide với 1 đầu DIG-11-ddUTP/ 1 phân tử oligonucleotide.

- Phản ứng gắn đuôi 3’: vị trí terminal transferase gắn thêm hỗn hợp nucleotide không đánh dấu và DIG-11-dUTP là đuôi chứa nhiều thành phần DIG-11-dUTP.
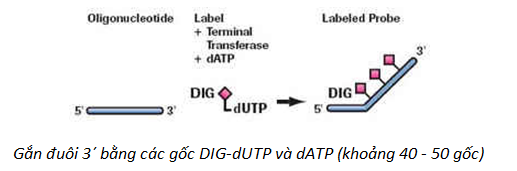
- Đánh dấu đầu 5’ gồm 2 bước: tổng hợp một cầu nối amino đầu 5’; sau khi tinh sạch, sau đó một digoxigenin-N-hydroxy-succinimide ester liên kết hóa trị với đầu 5'-amino tự do. Oligonucleotide cũng có thể được đánh dấu với bacteriophage T4 polynucleotide kinase. Nếu phản ứng thành công, hoạt động đặc hiệu của mẫu dò loại này có thể cao bằng hoạt động của [γ-32P] ATP.

Bài viết khác
TƯ VẤN VÀ XÂY DỰNG PHÒNG THÍ NGHIỆM AN TOÀN



